Vamos lembrar da curva Gaussiana/Normal que possui um formato de sino (figura 1). Ela não é muito alongada nas “pontas.” Ou seja, as observações não fogem muito da média. Quando usamos essa distribuição como verossimilhança na inferência modelos Bayesianos, forçamos a que todas as estimativas sejam condicionadas à uma distribuição normal da variável dependente. Se nos dados houverem muitas observações com valores discrepantes (bem diferentes da média – outliers), isso faz com que as estimativas dos coeficientes das variáveis independentes fiquem instáveis. Isso ocorre porquê a distribuição normal não consegue contemplar observações muito divergentes da média sem ter que mudar a média de posição. Em outras palavras, ela precisa se “deslocar” para conseguir estimar observações com valores discrepantes causando instabilidade da inferência.
ggplot(data = tibble(x = seq(-4, 4, length = 100))) +
labs(
title = "Distribuição Normais",
subtitle = expression(list(mu == 0, sigma == 1)),
x = expression(theta),
y = "Densidade"
) +
geom_line(aes(x, y = dnorm(x, mean = 0, sd = 1)), color = "steelblue", size = 2)
Figure 1: Distribuição Normal
Então precisamos de uma distribuição mais “maleável” como verossimilhança. Uma distribuição que seja mais robusta à observações discrepantes (outliers). Uma distribuição similar à Normal mas que possua caudas mais longas para justamente contemplar observações muito longe da média sem ter que mudar a média de posição e ter que se “contorser” toda. Para isso temos a distribuição \(t\) de Student. Lembrando o formato dela na figura 2:
ggplot(data = tibble(x = seq(-4, 4, length = 100))) +
labs(
title = "Distribuição t de Student",
subtitle = expression(list(nu == 3, mu == 0, sigma == 1)),
x = expression(theta),
y = "Densidade"
) +
geom_line(aes(x, y = dt(x, df = 3)), color = "red", size = 2)
Figure 2: Distribuição \(t\) de Student
Comparativo Normal vs Student
Reparem nas caudas na figura 3:
ggplot(data = tibble(x = seq(-4, 4, length = 100))) +
labs(
title = "Comparativo Distribuições Normal vs t de Student",
subtitle = expression(list(nu == 3, mu == 0, sigma == 1)),
x = expression(theta),
y = "Densidade"
) +
geom_line(aes(x, y = dnorm(x, mean = 0, sd = 1), color = "Normal"), size = 2) +
geom_line(aes(x, y = dt(x, df = 3), color = "Student"), size = 2) +
scale_color_manual(
name = "Distribuições",
values = c("steelblue", "red")
)
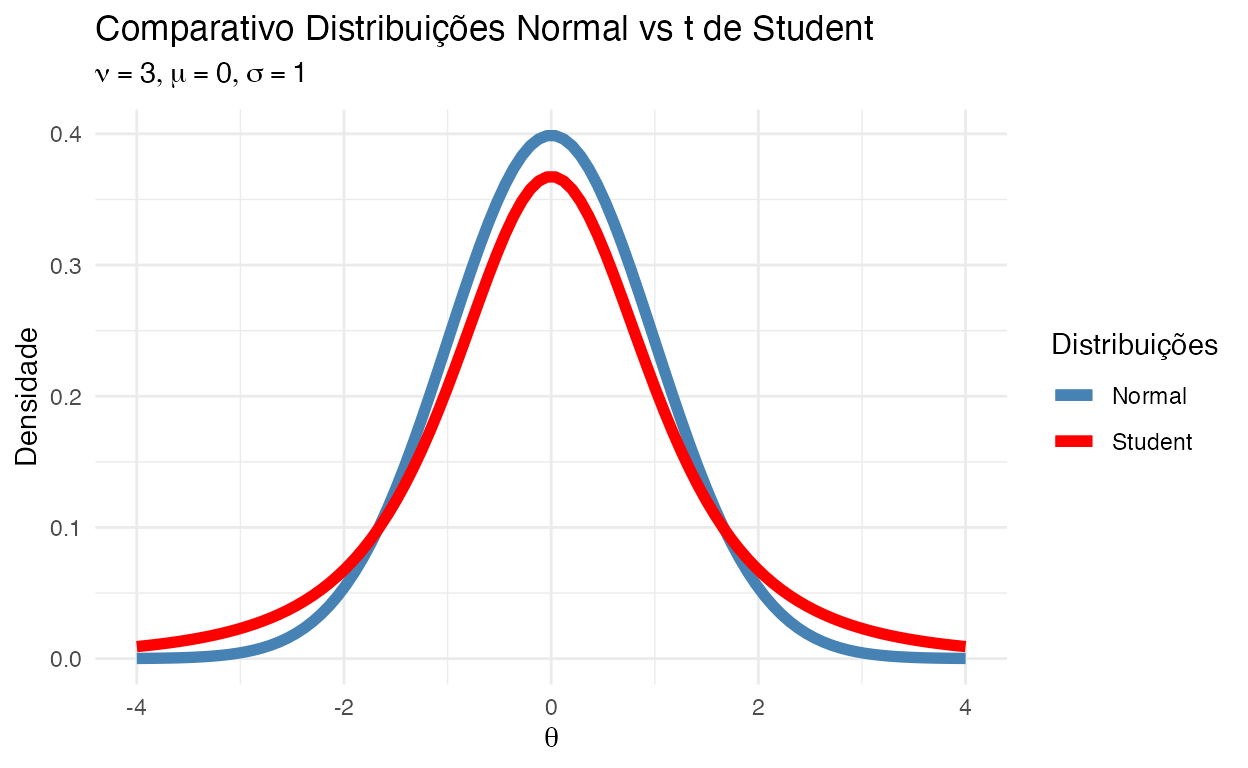
Figure 3: Comparativo Distribuições Normal vs \(t\) de Student
Regressão Robusta Bayesiana
A abordagem padrão para modelarmos uma variável dependente contínua é com um função de verossimilhança Gaussiana/Normal. Isto implica que o erro do modelo, \(\sigma\) da função de verossimilhança Gaussiana/Normal seja distribuído como uma distribuição normal:
\[ \begin{aligned} \boldsymbol{y} &\sim \text{Normal}\left( \alpha + \mathbf{X} \boldsymbol{\beta}, \sigma \right) \\ \alpha &\sim \text{Normal}(\mu_\alpha, \sigma_\alpha) \\ \boldsymbol{\beta} &\sim \text{Normal}(\mu_{\boldsymbol{\beta}}, \sigma_{\boldsymbol{\beta}}) \\ \sigma &\sim \text{Exponencial}(\lambda_\sigma) \end{aligned} \]
Do ponto de vista Bayesiano, não há nada especial na verossimilhança Gaussiana/Normal. É apenas uma distribuição probabilística especificada em um modelo. Podemos deixar o modelo mais robusto ao usarmos uma distribuição \(t\) de Student como função de verossimilhança. Isto implica que o erro do modelo, \(\sigma\) não segue uma distribuição normal, mas sim uma distribuição \(t\) de Student:
\[ \begin{aligned} \boldsymbol{y} &\sim \text{Student}\left( \nu, \alpha + \mathbf{X} \boldsymbol{\beta}, \sigma \right) \\ \alpha &\sim \text{Normal}(\mu_\alpha, \sigma_\alpha) \\ \boldsymbol{\beta} &\sim \text{Normal}(\mu_{\boldsymbol{\beta}}, \sigma_{\boldsymbol{\beta}}) \\ \nu &\sim \text{Log-Normal}(2, 1) \\ \sigma &\sim \text{Exponencial}(\lambda_\sigma) \end{aligned} \]
Aqui temos algumas diferenças:
- A função de verossimilhança \(t\) de Student requer 1 parâmetro adicional: \(\nu\), os graus de liberdade. Esses controlam o quão “longas” serão as caudas. Valores \(\nu > 20\) fazem a distribuição \(t\) de Student praticamente se tornar uma distribuição normal.
- Não há nada especial em \(\nu\). É apenas mais um parâmetro para o modelo estimar. Então é só eluciar uma priori. No caso, estou usando uma Distribuição Log-Normal com média 2 e desvio padrão 1.
Veja que não há nada de especial também nas prioris dos coeficientes \(\boldsymbol{\beta}\) ou da constante \(\alpha\). Poderíamos muito bem também colocar outras distribuições ou até deixar o modelo mais robustos ainda à outliers colocando prioris \(t\) de Student:
\[ \begin{aligned} \alpha &\sim \text{Student}(\nu_\alpha, \mu_\alpha, \sigma_\alpha) \\ \boldsymbol{\beta} &\sim \text{Student}(\nu_{\boldsymbol{\beta}}, \mu_{\boldsymbol{\beta}}, \sigma_{\boldsymbol{\beta}}) \\ \nu_\alpha &\sim \text{Log-Normal}(1, 1) \\ \nu_{\boldsymbol{\beta}} &\sim \text{Log-Normal}(1, 1) \end{aligned} \]
Modelos Lineares Robustos com o pacote brms
O rstanarm não nos dá a possibilidade de usar distribuições \(t\) de Student como função de verossimilhança de modelos Bayesiano. Para usarmos distribuições \(t\) de Student, precisamos usar o brms (Bürkner, 2017). O brms usa a mesma síntaxe que o rstanarm e possui as mesmas famílias de funções de verossimilhança, mas com algumas diferenças:
rstanarmtodos os modelos são pré-compilados ebrmsnão possui os modelos pré-compilados então os modelos devem ser todos compilados antes de serem rodados. A diferença prática é que você irá esperar alguns instantes antes do R começar a amostrar do modelo.- Como
brmscompila os modelos conforme a especificação do usuário (não possui modelos pré-compilados) ele pode criar modelos um pouco mais eficientes que orstanarm. Caso o tempo de compilação dos modelosbrmsseja menor que ganho de velocidade em amostragem do modelo, é vantajoso especificar seu modelo combrms. brmsdá maior poder e flexibilidade ao usuário na especificação de funções de verossimilhança e também permite modelos mais complexos que orstanarm.
A função que usamose para designar modelos Bayesianos no brms é a brm():
brm(y ~ ...,
data = ...,
family = student)
Exemplo - Prestígio de Duncan (1961)
Para exemplicar regressão robusta vamos usar um dataset que tem muitas observações discrepantes (outliers) chamado Duncan (Duncan, 1961). Ele possui 45 observações sobre ocupações nos EUA e 5 variáveis:
profession: Nome da profissãotype: Tipo de ocupação. Uma variável qualitativa:prof- profissional ou de gestãowc- white-collar (colarinho branco)bc- blue-collar (colarinho azul)
income: Porcentagem de pessoas da ocupação que ganham acima U$ 3.500 por ano em 1950 (mais ou menos U$36.000 em 2017);education: Porcentagem de pessoas da ocupação que possuem diploma de ensino médio em 1949 (que, sendo cínicos, podemos dizer que é de certa maneira equivalente com diploma de Doutorado em 2017); eprestige: Porcentagem de respondentes na pesquisa que classificam a sua ocupação como no mínimo “boa” em respeito à prestígio.
Como sempre eu gosto de usar o pacote skimr (Waring et al., 2021) com a função skim():
| Name | duncan |
| Number of rows | 45 |
| Number of columns | 5 |
| _______________________ | |
| Column type frequency: | |
| factor | 2 |
| numeric | 3 |
| ________________________ | |
| Group variables | None |
Variable type: factor
| skim_variable | n_missing | complete_rate | ordered | n_unique | top_counts |
|---|---|---|---|---|---|
| profession | 0 | 1 | FALSE | 45 | acc: 1, pil: 1, arc: 1, aut: 1 |
| type | 0 | 1 | FALSE | 3 | bc: 21, pro: 18, wc: 6 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| income | 0 | 1 | 42 | 24 | 7 | 21 | 42 | 64 | 81 | ▇▂▅▃▅ |
| education | 0 | 1 | 53 | 30 | 7 | 26 | 45 | 84 | 100 | ▆▆▃▂▇ |
| prestige | 0 | 1 | 48 | 32 | 3 | 16 | 41 | 81 | 97 | ▇▃▅▂▇ |
Eu acho que variável dependente prestige merece uma atenção maior, vejam o histograma dela na figura 4. Parece que temos uma multimodalidade rolando por aqui:
ggplot(duncan, aes(prestige)) +
geom_histogram(bins = 15, fill = "steelblue") +
labs(
title = "Histograma da variável prestige",
y = "Frequência"
)
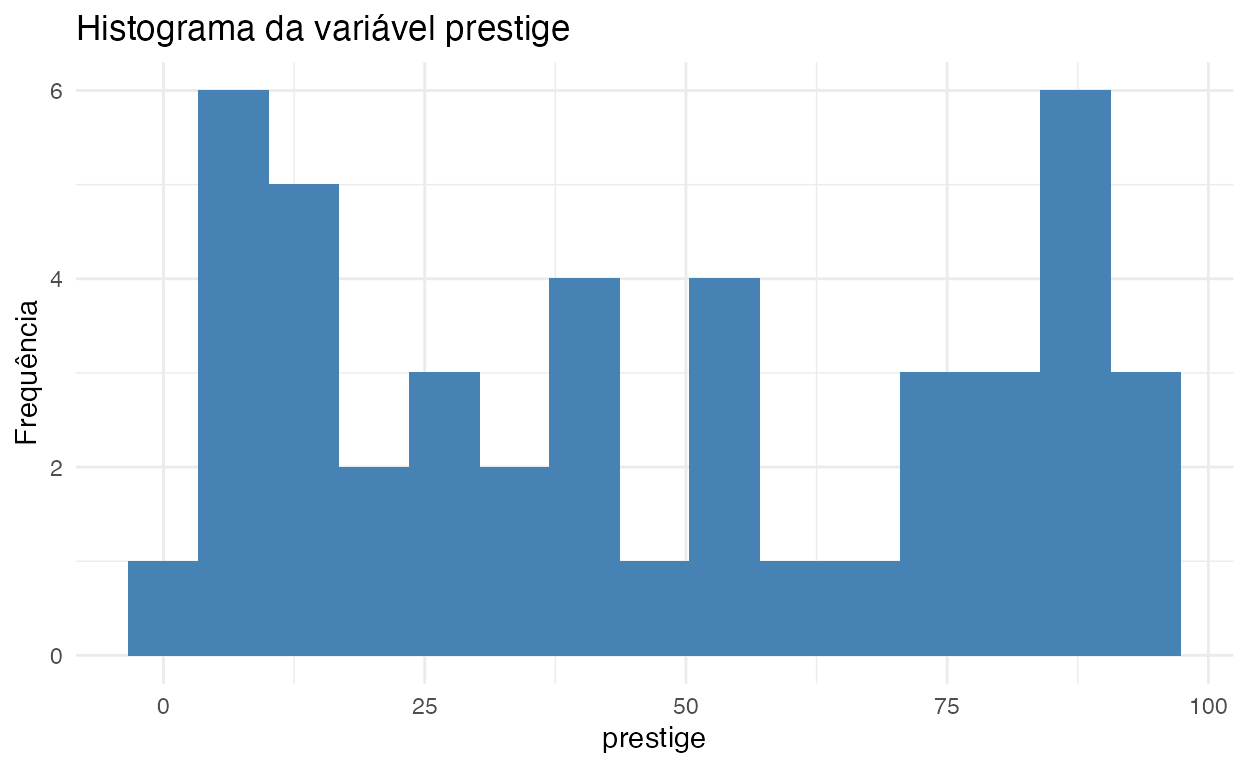
Figure 4: Histograma da variável prestige
Primeiro modelo: Regressão Linear
Vamos estimar primeiramente uma regressão linear usando a distribuição Normal como verossimilhança no rstanarm. Para exemplificação vou manter as prioris padrões do rstanarm:
# Detectar quantos cores/processadores
options(mc.cores = parallel::detectCores())
options(Ncpus = parallel::detectCores())
library(rstanarm)
model_1 <- stan_glm(
prestige ~ income + education,
data = duncan,
family = gaussian # nem precisaria pois é o argumento padrão
)
E na sequência o sumário das estimativas do modelo, assim como os diagnósticos da MCMC:
summary(model_1)
Model Info:
function: stan_glm
family: gaussian [identity]
formula: prestige ~ income + education
algorithm: sampling
sample: 4000 (posterior sample size)
priors: see help('prior_summary')
observations: 45
predictors: 3
Estimates:
mean sd 10% 50% 90%
(Intercept) -6.1 4.6 -12.0 -6.2 -0.2
income 0.6 0.1 0.4 0.6 0.8
education 0.5 0.1 0.4 0.5 0.7
sigma 13.8 1.6 11.8 13.6 15.8
Fit Diagnostics:
mean sd 10% 50% 90%
mean_PPD 47.6 2.9 43.9 47.7 51.3
The mean_ppd is the sample average posterior predictive distribution of the outcome variable (for details see help('summary.stanreg')).
MCMC diagnostics
mcse Rhat n_eff
(Intercept) 0.1 1.0 4499
income 0.0 1.0 2089
education 0.0 1.0 2319
sigma 0.0 1.0 1977
mean_PPD 0.1 1.0 3023
log-posterior 0.0 1.0 1343
For each parameter, mcse is Monte Carlo standard error, n_eff is a crude measure of effective sample size, and Rhat is the potential scale reduction factor on split chains (at convergence Rhat=1).Aparentemente parece que o modelo possui boas métricas (todos Rhat menores que 1.01) mas quando olhamos o Posterior Predictive Check (figura 5), vemos uma bagunça:
pp_check(model_1, nreps = 40)
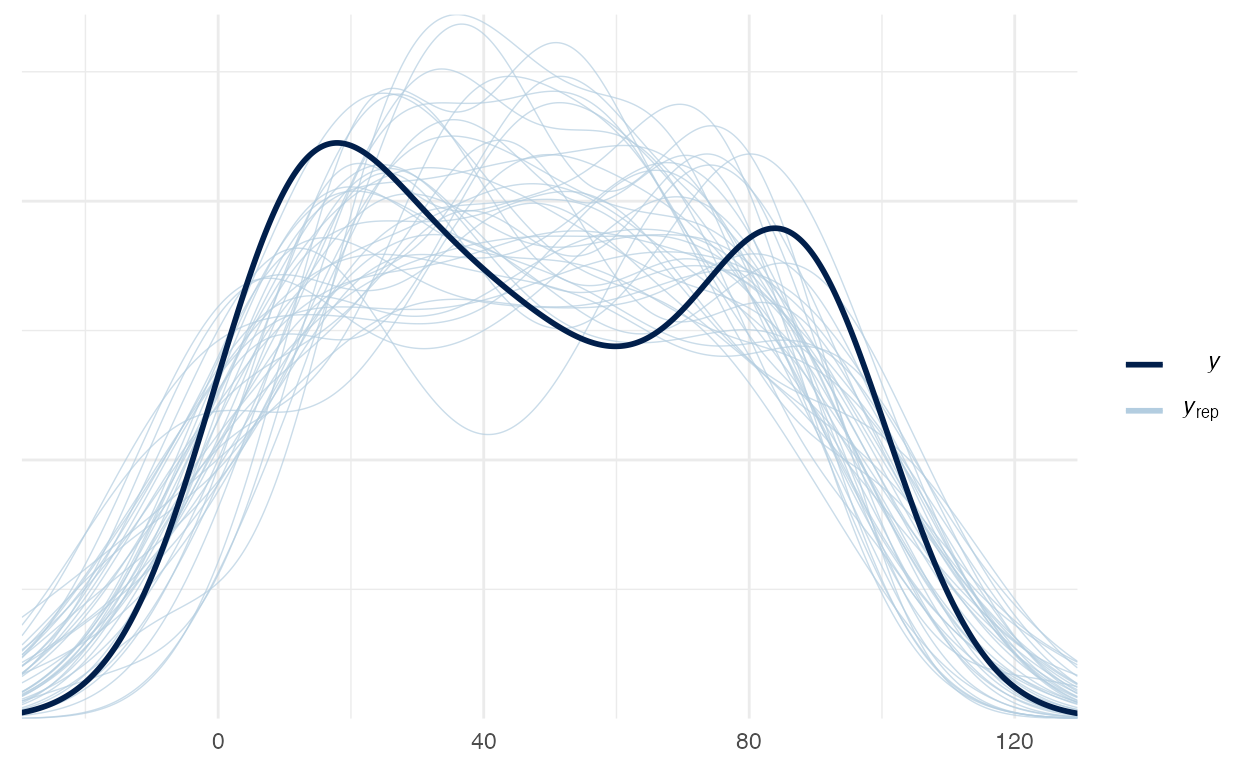
Figure 5: Posterior Preditive Check do modelo Gaussiano/Normal
Segundo modelo: Regressão Robusta
Para rodar um modelo Bayesiano que usa como verossimilhança a distribuição \(t\) de Student basta somente usar a mesma síntaxe que o stan_glm mas colocando argumento family = student. Aqui vou usar as mesmas priors que o padrão do rstanarm (caso queira, é só verificá-las com o prior_summary(model_1)):
library(brms)
model_2 <- brm(
prestige ~ income + education,
data = duncan,
family = student,
prior = c(
prior(normal(0, 3.22), class = b, coef = income),
prior(normal(0, 2.65), class = b, coef = education),
prior(normal(48, 79), class = Intercept),
prior(exponential(0.032), class = sigma),
prior(lognormal(2, 1), class = nu)
)
)
E na sequência o sumário das estimativas do modelo com verossimilhança \(t\) de Student (estou colocando o prob = 0.9 para que o sumário do modelo brms seja o mesmo que o sumário do rstanarm), assim como os diagnósticos da MCMC:
summary(model_2, prob = 0.9)
Family: student
Links: mu = identity; sigma = identity; nu = identity
Formula: prestige ~ income + education
Data: duncan (Number of observations: 45)
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup samples = 4000
Population-Level Effects:
Estimate Est.Error l-90% CI u-90% CI Rhat Bulk_ESS Tail_ESS
Intercept -7.03 3.91 -13.29 -0.40 1.00 4796 3249
income 0.69 0.14 0.46 0.91 1.00 1582 2198
education 0.49 0.11 0.32 0.67 1.00 1615 2057
Family Specific Parameters:
Estimate Est.Error l-90% CI u-90% CI Rhat Bulk_ESS Tail_ESS
sigma 11.27 2.13 7.67 14.72 1.00 1383 1501
nu 10.70 12.35 2.09 33.90 1.00 1676 1549
Samples were drawn using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).Vemos que o erro do modelo sigma reduziu de \(\approx 14\) para \(\approx 11\), o que implica em uma melhor inferência do modelo. Além disso, temos um novo parâmetro estimado pelo modelo que é o parâmetro nu (\(\nu\)): os graus de liberdade da distribuição \(t\) de Student usada como verossimilhança.
E para concluir, vejam que p Posterior Predictive Check (figura 6) ficou com um aspecto muito melhor que o modelo linear:
pp_check(model_2, nsamples = 40)
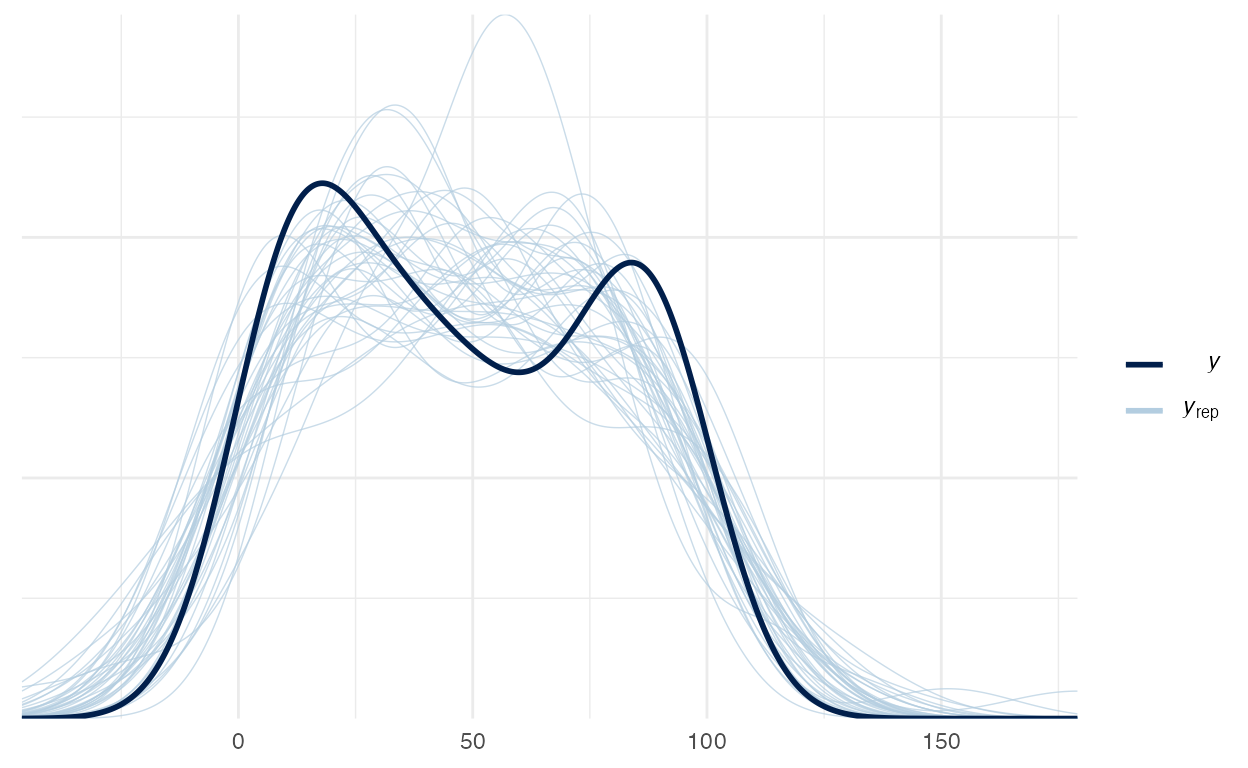
Figure 6: Posterior Preditive Check do modelo \(t\) de Student
Atividade Prática
O dataset Boston Housing está disponível em datasets/Boston_Housing.csv. Possui 506 observações e possui 14 variáveis:
CRIM- per capita crime rate by townZN- proportion of residential land zoned for lots over 25,000 sq.ft.INDUS- proportion of non-retail business acres per town.CHAS- Charles River dummy variable (1 if tract bounds river; 0 otherwise)NOX- nitric oxides concentration (parts per 10 million)RM- average number of rooms per dwellingAGE- proportion of owner-occupied units built prior to 1940DIS- weighted distances to five Boston employment centresRAD- index of accessibility to radial highwaysTAX- full-value property-tax rate per $10,000PTRATIO- pupil-teacher ratio by townB- 1000(Bk - 0.63)^2 where Bk is the proportion of blacks by townLSTAT- % lower status of the populationMEDV- Median value of owner-occupied homes in $1000’s
###
Ambiente
R version 4.1.0 (2021-05-18)
Platform: x86_64-apple-darwin17.0 (64-bit)
Running under: macOS Big Sur 10.16
Matrix products: default
BLAS: /Library/Frameworks/R.framework/Versions/4.1/Resources/lib/libRblas.dylib
LAPACK: /Library/Frameworks/R.framework/Versions/4.1/Resources/lib/libRlapack.dylib
locale:
[1] en_US.UTF-8/en_US.UTF-8/en_US.UTF-8/C/en_US.UTF-8/en_US.UTF-8
attached base packages:
[1] stats graphics grDevices utils datasets methods
[7] base
other attached packages:
[1] gapminder_0.3.0 ggExtra_0.9 dplyr_1.0.6
[4] rstan_2.21.2 StanHeaders_2.21.0-7 MASS_7.3-54
[7] ggforce_0.3.3 gganimate_1.0.7 plotly_4.9.3
[10] carData_3.0-4 DiagrammeR_1.0.6.1 brms_2.15.0
[13] rstanarm_2.21.1 Rcpp_1.0.6 skimr_2.1.3
[16] readr_1.4.0 readxl_1.3.1 tibble_3.1.2
[19] ggplot2_3.3.3 patchwork_1.1.1 cowplot_1.1.1
loaded via a namespace (and not attached):
[1] backports_1.2.1 systemfonts_1.0.2 plyr_1.8.6
[4] igraph_1.2.6 lazyeval_0.2.2 repr_1.1.3
[7] splines_4.1.0 crosstalk_1.1.1 rstantools_2.1.1
[10] inline_0.3.19 digest_0.6.27 htmltools_0.5.1.1
[13] magick_2.7.2 rsconnect_0.8.18 fansi_0.5.0
[16] magrittr_2.0.1 RcppParallel_5.1.4 matrixStats_0.59.0
[19] xts_0.12.1 prettyunits_1.1.1 colorspace_2.0-1
[22] textshaping_0.3.4 xfun_0.23 callr_3.7.0
[25] crayon_1.4.1 jsonlite_1.7.2 lme4_1.1-27
[28] survival_3.2-11 zoo_1.8-9 glue_1.4.2
[31] polyclip_1.10-0 gtable_0.3.0 V8_3.4.2
[34] pkgbuild_1.2.0 abind_1.4-5 scales_1.1.1
[37] mvtnorm_1.1-1 DBI_1.1.1 miniUI_0.1.1.1
[40] isoband_0.2.4 progress_1.2.2 viridisLite_0.4.0
[43] xtable_1.8-4 tmvnsim_1.0-2 stats4_4.1.0
[46] DT_0.18 httr_1.4.2 htmlwidgets_1.5.3
[49] threejs_0.3.3 RColorBrewer_1.1-2 ellipsis_0.3.2
[52] pkgconfig_2.0.3 loo_2.4.1 farver_2.1.0
[55] sass_0.4.0 here_1.0.1 utf8_1.2.1
[58] tidyselect_1.1.1 labeling_0.4.2 rlang_0.4.11
[61] reshape2_1.4.4 later_1.2.0 visNetwork_2.0.9
[64] munsell_0.5.0 cellranger_1.1.0 tools_4.1.0
[67] cli_2.5.0 generics_0.1.0 gifski_1.4.3-1
[70] ggridges_0.5.3 evaluate_0.14 stringr_1.4.0
[73] fastmap_1.1.0 yaml_2.2.1 ragg_1.1.2
[76] processx_3.5.2 knitr_1.33 purrr_0.3.4
[79] nlme_3.1-152 mime_0.10 projpred_2.0.2
[82] xml2_1.3.2 compiler_4.1.0 bayesplot_1.8.0
[85] shinythemes_1.2.0 rstudioapi_0.13 curl_4.3.1
[88] gamm4_0.2-6 png_0.1-7 tweenr_1.0.2
[91] bslib_0.2.5.1 stringi_1.6.2 highr_0.9
[94] ps_1.6.0 Brobdingnag_1.2-6 lattice_0.20-44
[97] Matrix_1.3-3 nloptr_1.2.2.2 markdown_1.1
[100] shinyjs_2.0.0 vctrs_0.3.8 pillar_1.6.1
[103] lifecycle_1.0.0 jquerylib_0.1.4 bridgesampling_1.1-2
[106] data.table_1.14.0 httpuv_1.6.1 R6_2.5.0
[109] bookdown_0.22 promises_1.2.0.1 gridExtra_2.3
[112] codetools_0.2-18 distill_1.2 boot_1.3-28
[115] colourpicker_1.1.0 gtools_3.8.2 assertthat_0.2.1
[118] rprojroot_2.0.2 withr_2.4.2 mnormt_2.0.2
[121] shinystan_2.5.0 mgcv_1.8-35 parallel_4.1.0
[124] hms_1.1.0 grid_4.1.0 tidyr_1.1.3
[127] coda_0.19-4 minqa_1.2.4 rmarkdown_2.8
[130] downlit_0.2.1 shiny_1.6.0 lubridate_1.7.10
[133] base64enc_0.1-3 dygraphs_1.1.1.6